Comparative analysis of intestinal expression of microRNAs in pediatric inflammatory bowel disease
Summary
Recently, special epigenetic mechanisms came into view with increasing interest toward the involvement of small non-coding RNAs called microRNAs (miR) that are able to regulate the gene expression. The field of miR research is rapidly expanding in adult inflammatory bowel disease (IBD), however, less is known about pediatric IBD. The aim of my PhD work was to characterize a pediatric IBD (IBD) miR profile that might serve as potential Crohn’s disease (CD) and ulcerative colitis (UC) specific diagnostic pattern. We demonstrated an IBD characteristic microRNA pattern by next-generation sequencing and RT-PCR that could facilitate deeper understanding of the pathomechanism of IBD. MiR-18a, -21, -31, 125a, -142-3p, -146a, -185 and -223 may serve as a diagnostic tool in the future. In addition, our next aim was to analyze the related target genes and their biological functions of miRs to reveal their complex role in the pathomechanism of IBD. The most important IBD-related functional groups and processes including inflammation, fibrosis and angiogenesis. Thus providing further evidence for the specificity of the miR profile and underlining the potential importance of these regulatory elements in the pathomechanism of IBD, they could serve as potential differential diagnostic markers and therapeutic targets.
Az utóbbi időben számos kórkép patomechanizmusának megértésében előtérbe kerültek a legújabb epigenetika tárgykörében végzett kutatások, köztük a rövid, nem kódoló mikroRNS-ek. A mikroRNS-ek fő funkciói közé tartozik a génexpresszió szabályozása. A mikroRNS-ekkel kapcsolatos kutatások száma gyorsan növekszik a felnőtt gyulladásos bélbetegségben (IBD), azonban gyermekkori indulású IBD-ben kevés adat áll rendelkezésünkre. Éppen ezért választottuk PhD munkám céljának az IBD-s gyermekek biopsziás mintáinak mikroRNS profil meghatározását újgenerációs szekvenálással, majd valós idejű polimeráz láncreakcióval (RT-PCR) történő analízisét. Eredményeink alapján a miR-18a, -21, -31, 125a, -142-3p, -146a, -185 és -223 potenciális diagnosztikai marker lehet a jövőben. Ezt követően az érintett mikroRNS-ek célgénjeinek és azok biológiai funkcióinak meghatározását végeztük el. A legfontosabb gyermekkori IBD-hez köthető funkciós csoportok, amelyeket az általunk vizsgált mikroRNS-ek regulálnak a gyulladásos folyamatok, a fibrózis és az angiogenezis szabályozása. Mindezek az eredmények tovább növelik az általunk vizsgált mikroRNS-ek jelentőségét a gyermekkori IBD patomechanizmusában, ezáltal potenciális terápiás célpontok és differenciáldiagnosztikai markerek kerülhetnek előtérbe.
Bevezetés
A gyulladásos bélbetegség (inflammatory bowel disease, IBD) a gasztrointesztinális traktus remissziókkal és relapszusokkal jellemezhető krónikus gyulladással járó megbetegedései. Két fő formája a Crohn-betegség (CD) és a colitis ulcerosa (UC), amelyek mellett megkülönböztetjük a két csoportba egyértelműen nem besorolható eseteket (IBD-unclassified, IBD-U) (1, 2). Az IBD 15-30%-ban gyermekkorban kezdődik. A koraibb betegségindulás hosszabb betegséglefolyást jelent és így várhatóan több a komplikáció. A felnőtt és gyermekkori IBD mind aktivitásában, mind lokalizációjában számos különbséget mutat (3–5). Az IBD multifaktoriális kórkép, kialakulásában genetikai, epigenetikai, immunológiai és környezeti tényezők egyaránt szerepet játszanak, azonban pontos oka máig sem tisztázott (6).
Néhány évtizede még az egyedi tulajdonságok-, illetve a fenotípus kialakulását az akkori tudományos kutatások alapján a DNS® RNS® fehérjelánc alapján determinálták. A „Human Genom Project” során azonban kiderült, hogy génállományunk csupán igen kis része kódol fehérjéket. Az epigenetika a genetikai és a környezeti tényezők közti kapocsként olyan jelenségekkel foglalkozik, amelyek következtében anélkül nő a genom genetikai sokféleséget létrehozó képessége, hogy a DNS szekvenciája változna. A legismertebb példa az Aegouti-gén metilációs vizsgálata, amelyben a genetikusan identikus egerek az anyai étrendtől függően különböző fenotípusúak lesznek (7). A legfontosabb epigenetikai mechanizmusok: a hiszton-modifikáció, a DNS-metiláció és a nem kódoló RNS-ek
(1. ábra).
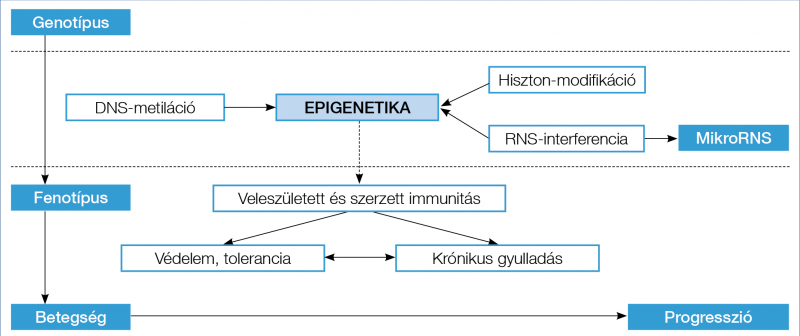
RNS-interferencia
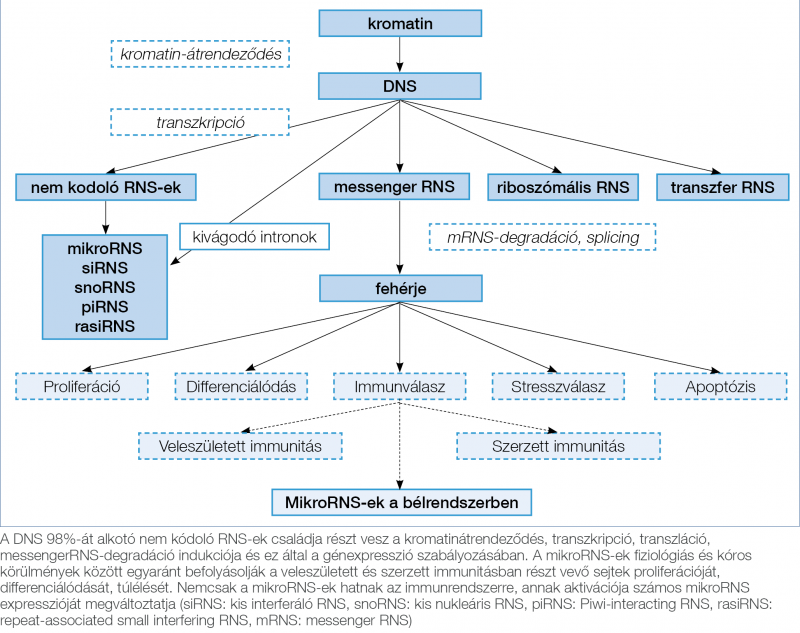
A nem kódoló RNS-ek, amelyek a fehérje-expresszió szabályozásában vesznek részt, méretük és funkciójuk szerint csoportosíthatóak (2. ábra). Jelenleg kb. 10 000 rövid, nem kódoló RNS-t ismerünk, amely a teljes genom (kb. 60 500 gén) csaknem 40%-át teszi ki (8, 9).
A mikroRNS-ek rövid, 18-24 nukleotidból álló, kétszálú RNS-ek, amelyek poszttranszkripciós szinten szabályozzák a génexpressziót (10). A miR-eket 1993-ban írták le a Caenorhabditis elegans (nematoda) vizsgálatakor a LIN-14 proteint kódoló lin-4 génnel kapcsolatban (11). A téma iránti fokozottan növekvő figyelmet jelzi, hogy míg 2000-ben összesen 3 közlemény jelent meg, addig 2018-ra ez a szám több ezerre emelkedett. A közelmúlt vizsgálatai egyértelművé tették, hogy ezen epigenetikai tényezőknek (mikroRNS, metiláció, hiszton-modifikáció) kiterjedt vizsgálata kulcsfontosságú lehet számos kórkép, így az IBD patomechanizmusában.
A mikroRNS-ek funkciója
Az elmúlt néhány év intenzív kutatásának köszönhetően kiderült, hogy több ezer mikroRNS létezik, és ezek szerepet játszanak a biológiai folyamatok regulálásában, valamint számos kórkép patomechanizmusában. A mikroRNS-ek az RNS-interferencia, géncsendesítés endogén mediátorai, poszttranszkripciós szinten szabályozzák a génexpressziót, valamint mRNS-bontó endonukleázok aktiválása révén mRNS bontást indukálnak (2. ábra) (8).
Egy sejttípuson belül minden mikroRNS-nek több száz mRNS célpontja lehet, illetve egy mRNS-hez akár egy időben is, több mikroRNS kapcsolódhat. Ennek megfelelően a humán transzkriptom nagy része a mikroRNS-ek regulációja alatt áll (10, 12).
Szerepet játszanak a differenciálódásban, a proliferáció- és az apoptózis szabályozásában, valamint a stressz- és immunválasz kialakításában (2. ábra). Ezen ismert funkcióik alapján eddig leginkább daganatos és immunmediált betegségekben vizsgálták szerepüket. Ezen túlmenően egyes mikroRNS-ek szerepet játszhatnak a kardiovaszkuláris-, neurodegeneratív, infekciós és metabolikus kórképek kialakulásában is (9, 10, 13, 14).
A jelenlegi kutatások fő iránya az egyes betegségekre specifikus mikroRNS-mintázatok meghatározása és funkciójuk azonosítása, amelyekkel lehetőség nyílik új diagnosztikus és prognosztikus markerek bevezetésére. Felmerül továbbá, hogy az adekvát mikroRNS-kezelés a jövőben terápiás értékűvé válik. A mikroRNS-ek terápiás felhasználásának két fő iránya:
1. a mikroRNS antagonisták,
2. mimikek használata.
MikroRNS-antagonisták
Fiziológiás körülmények között a mikroRNS-ek megnövekedett mennyisége csökkenti a célgének mennyiségét. Amennyiben a célgénnek védő funkciója van, a mikroRNS „overexpressziója” miatt kialakuló csökkenő célgén mennyiséget mikroRNS-antagonistákkal előzhetjük meg. A mikroRNS-antagonisták az eredeti mikroRNS-komplementer nukleotidjai, amelyeket különböző nukleázoknak ellenálló struktúrákba helyeznek. Jelenleg a miR-122 antagonistája 2. fázis vizsgálatban (Miravirsen®) van a hepatitis C-vírus (HCV) fertőzés terápiájában (15).
MikroRNS mimik-en alapuló terápia
Amikor egy mikroRNS-nek csökkent a szintje, a proinflammatórikus célgének felszabadulnak a gátlás alól, a gyulladásos folyamatok kerülnek előtérbe. Ennek terápiás megoldása lehet a mikroRNS mimik-ek alkalmazása, azaz a sejten belüli mikroRNS-pótlás, ezáltal a célgén mennyiségének csökkenése. A mikroRNS mimik-ek szintetikus RNS-szálak, amelyek kémiai módosításokkal válnak stabil formává. Különböző onkogének gátló mikroRNS-ei szintén terápiás célpontok, amelyek számos preklinikai vizsgálat részét képezik (16).
MikroRNS-ek a bélrendszerben
A bélrendszer baktériumai komplex módon szabályozott szimbiotikus kapcsolatban vannak a mucosat alkotó epithel- és immunsejtekkel. A bélnyálkahártya-egyensúly felborulása számos gyulladásos kórkép kialakulásához vezethet. A mikroRNS-ek számos, az immunrendszerrel, gyulladással összefüggő szignál transzdukciós útvonalat és génexpressziót regulálnak a gazdaszervezetben, amelyeknek fontos szerepe van a bélrendszerben végbemenő folyamatokban. A gazdaszervezet és a kommenzális baktériumok szimbiózisának megőrzésében az utóbbi években a mikroRNS-ek fontos szabályozó szerepét azonosították. Befolyásolják a baktériumok felismerését szabályozó gének expresszióját és az immunrendszer válaszának csökkentését, ezáltal szerepük van a tolerancia kialakulásában (17).
MikroRNS-ek IBD-ben
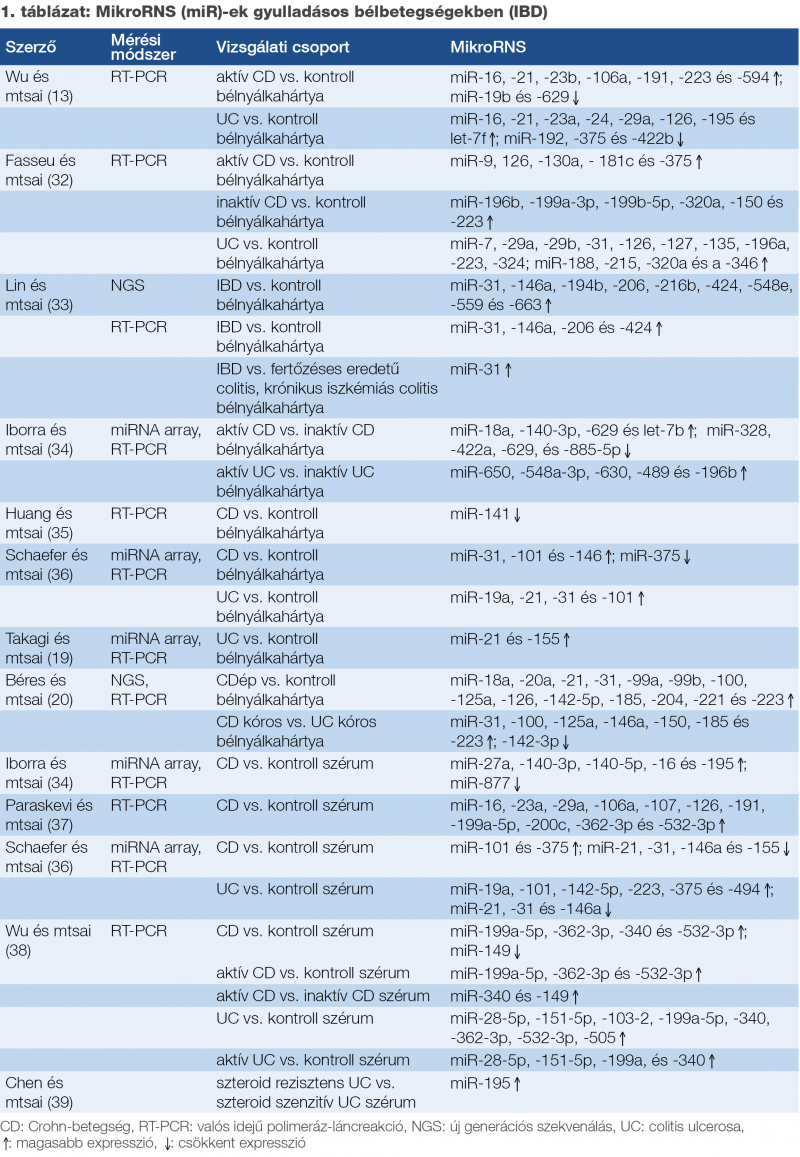
Az IBD-vel foglalkozó kutatások célja a betegség patomechanizmusának a pontosabb megértése, illetve non invazív diagnosztikai markerek keresése. 2008-ban Wu és munkatársai elsőként végeztek IBD-s betegek biopsziás mintáin. Munkájuk során aktív és inaktív UC-s, aktív CD-s, irritábilis bélszindrómás, mikroszkopikus colitises, fertőzéses colitises és kontroll felnőttek szigma biopsziáit vizsgálták. A mikroRNS-expresszió a CD (terminalis ileum és vastagbél-érintettség) csoportok között nem mutatott eltérést, azonban a kontrollcsoporthoz képest emelkedett expressziót mutatott a miR-16, -21, -23b, -106a, -191, -223 és -594, míg a miR-19b és -629 szintje csökkent. Emellett emelkedett miR-199a-5p, -362-3p, -340 és -532-3p, és csökkent miR-149 szintet találtak CD-s betegek szérumában a kontrollcsoporthoz képest (18). Takagi és munkatársai igazolták a miR-21 és -155 expresszió emelkedését UC-s biopsziákban (19). Felnőttekben végzett további vizsgálatokat a nemzetközi irodalomból összegyűjtve az 1. táblázatban tüntettem fel.
MikroRNS-ek a gyermekkori IBD-ben
A mikroRNS-ek gyermekkori IBD-ben betöltött szerepével kapcsolatban azonban jelenleg nagyon kevés adat áll rendelkezésre, ezért munkacsoportunk mikroRNS-vizsgálatokat végzett IBD-s gyermekek biopsziás mintáin. Ehhez elsőként a nemzetközi irodalomnak megfelelően kisebb elemszámon új generációs szekvenálással meghatároztuk a potenciálisan érintett mikroRNS-ek körét. Az így kapott adatokat tovább elemeztük, kiválasztottunk további 18 mikroRNS-t validálásra, nagyobb elemszámon. Az érintett mikroRNS-ek és célgénjeik biológiai funkcióit meghatároztuk nemzetközi, nyilvános adatbázisok segítségével (20, 21).
Munkacsoportunk 170 eltérően diszregulált mikroRNS-t igazolt új generációs szekvenálással gyermkkori IBD-ben. Ezek közül 148 eltért a makroszkóposan gyulladt CD-s mintákban a makroszkóposan ép CD és kontroll mucosahoz képest. Ezen belül 114 mikroRNS diszregulált a gyulladt CD-s nyálkahártyában a kontrollcsoporthoz képest, míg 99 diszregulált az ép CD-mintákhoz képest. Összesen 61 mikroRNS mutatott eltérő expressziót a makroszkóposan ép CD biopsziákban a kontrollcsoporthoz képest, ezek között 22 olyan mikroRNS-t azonosítottunk, amelyek csak ebben a csoportban mutattak eltérő expressziót.
Makroszkóposan kóros nyálkahártyák eldifferenciálása
A miR-31, -125a, -146a és -223 szintje emelkedett a gyulladt régióból származó UC-s biopsziákban a makroszkóposan kóros CD-s mintákhoz képest. Ezzel ellentétben a miR-142-3p szintje a gyulladt régiókból származó CD-s biopsziákban emelkedett az UC-s mintákhoz képest (3. ábra). A mikroRNS-ek diagnosztikai hatékonyságának vizsgálatára ROC (Receiver Operating Characteristic) analízist végeztünk, amelyben a görbe alatti terület (AUC) a hatékonyság erősségét jelző érték, ahol a célérték: 1. A ROC-analízis alapján a miR-142-3p (AUC: 0,888 p<0,01; szenzitivitás 77,78%, specificitás: 90,31%) és -146a (AUC:0,838 p<0,01; szenzitivitás 80%, specificitás: 76,92%) magas szenzitivitással és specificitással képes elkülöníteni a két kórképet. Mindezek alapján a miR-31, -125a, -142-3p, -146a és -223 CD specifikus markerek lehetnek, amik segíthetnek a gyermekkori CD és UC differenciálásában. Eredményeink jelentőségét az adja, hogy pancolitis esetén sokszor nehézségekbe ütközik a CD és UC egymástól történő differenciálása. Gyermekkori UC-ben az esetek 55-75%-a kezdődik extenzív colitissel (22–24), továbbá számos pancolitises esetről számol be az irodalom CD-s gyermekek esetén (25). A hazai gyermekkori IBD-regiszter adatai alapján (Hungarian Pediatric IBD Registry Group, HUPIR) CD-sek kb. 4-6%-nál jelentkezik pancolitis, terminalis ileum érintettség és granulomák nélkül, amely megnehezíti a diagnózis felállítását. Mindezeknek terápiás konzekvenciája van, mivel egy UC-s gyermek pancolitise esetén szóba jön a colectomia, míg CD diagnózisánál igyekszünk a műtétet elkerülni. Éppen ezért különösen fontos CD és UC elkülönítését segítő biomarkerek azonosítása.

(CD ép) és kóros (CD kóros) CD, UC és kontroll vastagbél-biopsziákban
Makroszkóposan ép nyálkahártya elkülönítése
A miR-18a, -21, -31, 125a, -185 és -223 expressziója emelkedett a makroszkóposan ép CD-s biopsziás mintákban a kontrollcsoporthoz képest, ezzel ellentétben a miR-142-3p expressziója csökkent a CD-s gyermekek ép nyálkahártyájában a kontrollcsoporthoz képest (3. ábra). A ROC-analízis alapján a miR-185 (AUC: 0,81 p<0,05; szenzitivitás 62,5%, specificitás: 100%) és a miR-223 (AUC:1 p<0,001; szenzitivitás 100%, specificitás: 100%) magas szenzitivitással és specificitással képes különbséget tenni a makroszkóposan ép CD és kontroll nyálkahártya között. A CD diagnózisa nehezített izolált ilealis érintettség esetén, amikor az alsó és felső GI-traktus ép, az alsó- és felső endoszkópián nem látunk eltérést, a terminalis ileumba való bejutás nehézségbe ütközik technikai okból. Éppen ezért fontos lenne olyan biomarkerek azonosítása, amely ép vastagbél-nyálkahártya esetén is képes meghatározni a CD diagnózisát. Izolált ilealis érintettség 0,4-7%-ban fordulhat elő gyermekkori CD-ben (6, 22, 26). Továbbá az EUROKIDS-regiszter alapján az izolált terminalis ileum érintettség (± limitált coecum érintettség) a gyermekek 16%-ában fordul elő (27). Amennyiben a terminalis ileum intubációja nem jár sikerrel, ezekben az esetekben a diagnózis felállításához kapszulás endoszkópia, vagy altatásban végezhető MR-vizsgálat szükséges (28).
A mikroRNS-ek biológiai folyamatokban betöltött szerepének vizsgálata
A szekvenálással igazolt, IBD-ben releváns mikroRNS-ek által szabályozott célgéneket (MirTarBase, http://mirtarbase.mbc.nctu.edu.tw) (29) összehasonlítottuk nyilvános adatbázisból elérhető CD-s gyermekek biopsziáiból készült teljes transzkriptom szekvenálással kapott IBD-ben releváns génekkel. Az így kapott közös génhalmazt biológiai funkciók szerint (GO term-ek) csoportosítottuk. A transzkriptom szekvenálás (E-GEOD-57945) (30) és a mikroRNS-szekvenálás célgénjei alapján 126 gént vizsgáltunk tovább. Ezek a gének 248 biológiai folyamatban vesznek részt, amelyeket további 50 csoportba soroltunk összetartozásuk alapján. A csoportok 12 fő kategóriába oszthatók. A legjelentősebb csoportok: az apoptózis szabályozása, a hegképződésre adott válasz, a baktériumok elleni védekezés, a sejtmigráció és -adhézió, az érújdonképződés, a génexpresszió szabályozása, valamint a sejt-sejt jelátviteli utak szabályozása. Mindezek a biológiai feladatok fontosak az IBD patomechanizmusában, így ezek az eredmények tovább növelik az általunk vizsgált mikroRNS-ek jelentőségét a gyermekkori IBD-ben. Az általunk validált mikroRNS-ek adatai alapján számos olyan célgén is előtérbe került, amelyek pontos feladata még nem ismert az IBD patomechanizmusában, ezek potenciális terápiás célpontok lehetnek.
MikroRNS-ek, mint noninvazív diagnosztikus markerek
Noninvazív biomarkereket kutatva számos kórképben vizsgálták a különböző testnedvek mikroRNS expresszióját. Schaefer és munkatársai nyálban, szérumban és bélnyálkahártyán végeztek összehasonlító elemzéseket. Jelenleg kevés adat áll rendelkezésre a széklet mikroRNS expresszióra vonatkozóan IBD-s betegekben. Ahmed és munkatársai 5 felülexpresszált (miR-21, miR-203, miR-126 és miR-16) és 2 csökkent expressziót (miR-320 és -192) mutató mikroRNS-t azonosítottak UC-s betegek székletéből a kontrollcsoporthoz képest. A vizsgálat tárgyát képezte, hogy olyan mikroRNS-eket azonosítsanak, amelyek segítenek az UC és CRC azonosításában (31). A legfontosabb IBD-vel kapcsolatos mikroRNS-vizsgálatokat az 1. táblázat foglalja össze.
Következtetések
Az epigenetika tárgykörén belül az utóbbi években egyre több közlemény foglalkozik a mikroRNS-ek finomszabályozó szerepével a különböző gasztrointesztinális kórképek esetén. Mindezen eredmények alapján az ismerten diszregulált mikroRNS-ek közelebb vihetnek az egyes kórképek patomechanizmusának pontosabb megértéséhez, differenciáldiagnosztikai markerek és potenciális terápiás célpontok kerülhetnek előtérbe. A cél pedig különböző antiinflammatórikus és antifibrotikus készítmények fejlesztése.
2. Kovács M, Müller KE, Müller KE Fau, Papp M, Papp M, Lakatos PL, et al. New serological markers in pediatric patients with inflammatory bowel disease.
3. Baldassano RN, Piccoli DA. Inflammatory bowel disease in pediatric and adolescent patients. Gastroenterol Clin North Am 1999; 28: 445–458.
4. Kim SC, Ferry GD. Inflammatory bowel diseases in pediatric and adolescent patients: clinical, therapeutic, and psychosocial considerations. Gastroenterology 2004; 126: 1550–1560.
5. Perrin JM, Kuhlthau K, Chughtai A, et al. Measuring quality of life in pediatric patients with inflammatory bowel disease: psychometric and clinical characteristics. Journal of pediatric gastroenterology and nutrition 2008; 46: 164–171.
6. Muller KE, Lakatos PL, Arato A, et al. Incidence, Paris Classification, and Follow-up in a Nationwide Incident Cohort of Pediatric Patients With Inflammatory Bowel Disease. Journal of pediatric gastroenterology and nutrition 2013; 57: 576–582.
7. Chong SY, Vickaryous N, Ashe A, et al. Modifiers of epigenetic reprogramming show paternal effects in the mouse. Nature genetics 2007; 39: 614–622.
8. Mattick JS. Makunin IV. Non-coding RNA. Human molecular genetics. 2006; 15 Spec No1: R17–29.
9. Bhartiya D, Scaria V. Genomic variations in non-coding RNAs: Structure, function and regulation. LID – S0888-7543(16)30005-2 [pii] LID – 10.1016/j.ygeno.2016.01.005 [doi].
10. Chapman CG, Pekow J. The emerging role of miRNAs in inflammatory bowel disease: a review. Therapeutic advances in gastroenterology 2015; 8: 4–22.
11. Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell 1993; 75: 843–854.
12. Sevignani C, Calin GA, Siracusa LD, et al. Mammalian microRNAs: a small world for fine-tuning gene expression. Mammalian genome: official journal of the International Mammalian Genome Society 2006; 17: 189–202.
13. Wu F, Zikusoka M, Trindade A, et al. MicroRNAs are differentially expressed in ulcerative colitis and alter expression of macrophage inflammatory peptide-2 alpha. Gastroenterology 2008; 135: 1624–1635 e1624.
14. Dai R, Ahmed SA. MicroRNA, a new paradigm for understanding immunoregulation, inflammation, and autoimmune diseases. Translational research: the journal of laboratory and clinical medicine 2011; 157: 163–179.
15. Ottosen S, Parsley TB, Yang L, et al. In vitro antiviral activity and preclinical and clinical resistance profile of miravirsen, a novel anti-hepatitis C virus therapeutic targeting the human factor miR-122. Antimicrobial agents and chemotherapy 2015; 59: 599–608.
16. Reid G, Kao SC, Pavlakis N, et al. Clinical development of TargomiRs, a miRNA mimic-based treatment for patients with recurrent thoracic cancer. Epigenomics 2016; 8: 1079–1085.
17. Runtsch MC, Round JL, O’Connell RM. MicroRNAs and the regulation of intestinal homeostasis. Frontiers in genetics 2014; 5: 347.
18. Wu F, Zhang S, Dassopoulos T, et al. Identification of microRNAs associated with ileal and colonic Crohn’s disease. Inflammatory bowel diseases 2010; 16: 1729–1738.
19. Takagi T, Naito Y, Mizushima K, et al. Increased expression of microRNA in the inflamed colonic mucosa of patients with active ulcerative colitis. Journal of gastroenterology and hepatology 2010; 25(Suppl 1): S129–133.
20. Beres NJ, Kiss Z, Sztupinszki Z, et al. Altered mucosal expression of microRNAs in pediatric patients with inflammatory bowel disease. LID – S1590-8658(16)30861-1 [pii] LID – 10.1016/j.dld.2016.12.022 [doi].
21. Beres NJ, Szabo D, Kocsis D, et al. Role of Altered Expression of miR-146a, miR-155, and miR-122 in Pediatric Patients with Inflammatory Bowel Disease. Inflammatory bowel diseases 2016; 22: 327–335.
22. Van Limbergen J, Russell RK, Drummond HE, et al. Definition of phenotypic characteristics of childhood-onset inflammatory bowel disease. Gastroenterology 2008; 135: 1114–1122.
23. Muller KE, Lakatos PL, Kovacs JB, et al. Baseline Characteristics and Disease Phenotype in Inflammatory Bowel Disease. Journal of pediatric gastroenterology and nutrition 2016; 62: 50–55.
24. Levine A, de Bie CI, Turner D, et al. Atypical disease phenotypes in pediatric ulcerative colitis: 5-year analyses of the EUROKIDS Registry. Inflammatory bowel diseases 2013; 19: 370–377.
25. Lemberg DA, Clarkson CM, Bohane TD, et al. Role of esophagogastroduodenoscopy in the initial assessment of children with inflammatory bowel disease. Journal of gastroenterology and hepatology. 2005; 20: 1696–1700.
26. Meinzer U, Idestrom M, Alberti C, et al. Ileal involvement is age dependent in pediatric Crohn’s disease. Inflammatory bowel diseases 2005; 11: 639–644.
27. de Bie CI, Paerregaard A Fau – Kolacek S, Kolacek S Fau – Ruemmele FM, et al. Disease phenotype at diagnosis in pediatric Crohn’s disease: 5-year analyses of the EUROKIDS Registry.
28. Aloi M, Di Nardo G, Romano G, et al. Magnetic resonance enterography, small-intestine contrast US, and capsule endoscopy to evaluate the small bowel in pediatric Crohn’s disease: a prospective, blinded, comparison study. Gastrointest Endosc 2015; 81: 420–427.
29. Hsu SD, Tseng YT, Shrestha S, et al. miRTarBase update 2014: an information resource for experimentally validated miRNA-target interactions. Nucleic acids research 2014; 42: D78–85.
30. Haberman Y Fau – Tickle TL, Tickle Tl Fau – Dexheimer PJ, Dexheimer Pj Fau – Kim MO, et al. Pediatric Crohn disease patients exhibit specific ileal transcriptome and microbiome signature.
31. Ahmed FE, Jeffries CD, Vos PW, et al. Diagnostic microRNA markers for screening sporadic human colon cancer and active ulcerative colitis in stool and tissue. Cancer genomics & proteomics 2009; 6: 281–295.
32. Fasseu M, Treton X, Guichard C, et al. Identification of restricted subsets of mature microRNA abnormally expressed in inactive colonic mucosa of patients with inflammatory bowel disease. PloS one 2010; 5.
33. Lin J, Welker NC, Zhao Z, et al. Novel specific microRNA biomarkers in idiopathic inflammatory bowel disease unrelated to disease activity. Modern pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 2014; 27: 602–608.
34. Iborra M, Bernuzzi F, Correale C, et al. Identification of serum and tissue micro-RNA expression profiles in different stages of inflammatory bowel disease. Clinical and experimental immunology 2013; 173: 250–258.
35. Huang Z, Shi T, Zhou Q, et al. miR-141 Regulates colonic leukocytic trafficking by targeting CXCL12beta during murine colitis and human Crohn’s disease. Gut 2014; 63: 1247–1257.
36. Schaefer JS, Attumi T, Opekun AR, et al. MicroRNA signatures differentiate Crohn’s disease from ulcerative colitis. BMC Immunol 2015; 16: 5.
37. Paraskevi A, Theodoropoulos G, Papaconstantinou I, et al. Circulating MicroRNA in inflammatory bowel disease. Journal of Crohn’s & colitis 2012; 6: 900–904.
38. Wu F, Guo NJ, Tian H, et al. Peripheral blood microRNAs distinguish active ulcerative colitis and Crohn’s disease. Inflammatory bowel diseases 2011; 17: 241–250.
39. Chen G, Cao S, Liu F, et al. miR-195 plays a role in steroid resistance of ulcerative colitis by targeting Smad7. The Biochemical journal 2015; 471: 357–367.
